网站导航
医学图像分割 TGDAUNet算法简介
现在主流的医学图像分割方法主要是基于经典方法及深度学习。
而卷积神经网络CNN的出现加速了图像处理领域的发展。
基于CNN开发的GoogleNet7、ResNet8等网络模型在图像处理领域广泛应用, 但直至全卷积神经网络FCN的出现才加速了图像分割领域的研究。
U-Net发表于2015年,属于FCN的一种变体,是最先利用卷积神经网络进行医学影像分割的方法之一,分割的整体流程完全基于CNN。
U-Net跟FCN都是Encoder-Decoder结构,结构简单但很有效。
由于在医学方面,样本收集较为困难,为了解决这个问题,应用了图像增强的方法,在数据集有限的情况下获得了不错的精度。
U-Net 基础算法
U-Net网络结构是对称的,形似英文字母U所以被称为U-Net。 整张图都是由蓝/白色框与各种颜色的箭头组成,其中,蓝/白色框表示featuremap;蓝色箭头表示3x3卷积,用于特征提取;灰色箭头表示skip-connection, 用于特征融合;红色箭头表示池化pooling,用于降低维度;绿色箭头表示上采样upsample,用于恢复维度; 青色箭头表示1x1卷积,用于输出结果。其中灰色箭头copyandcrop中的copy就是concatenate而crop是为了让两者的长宽一致。
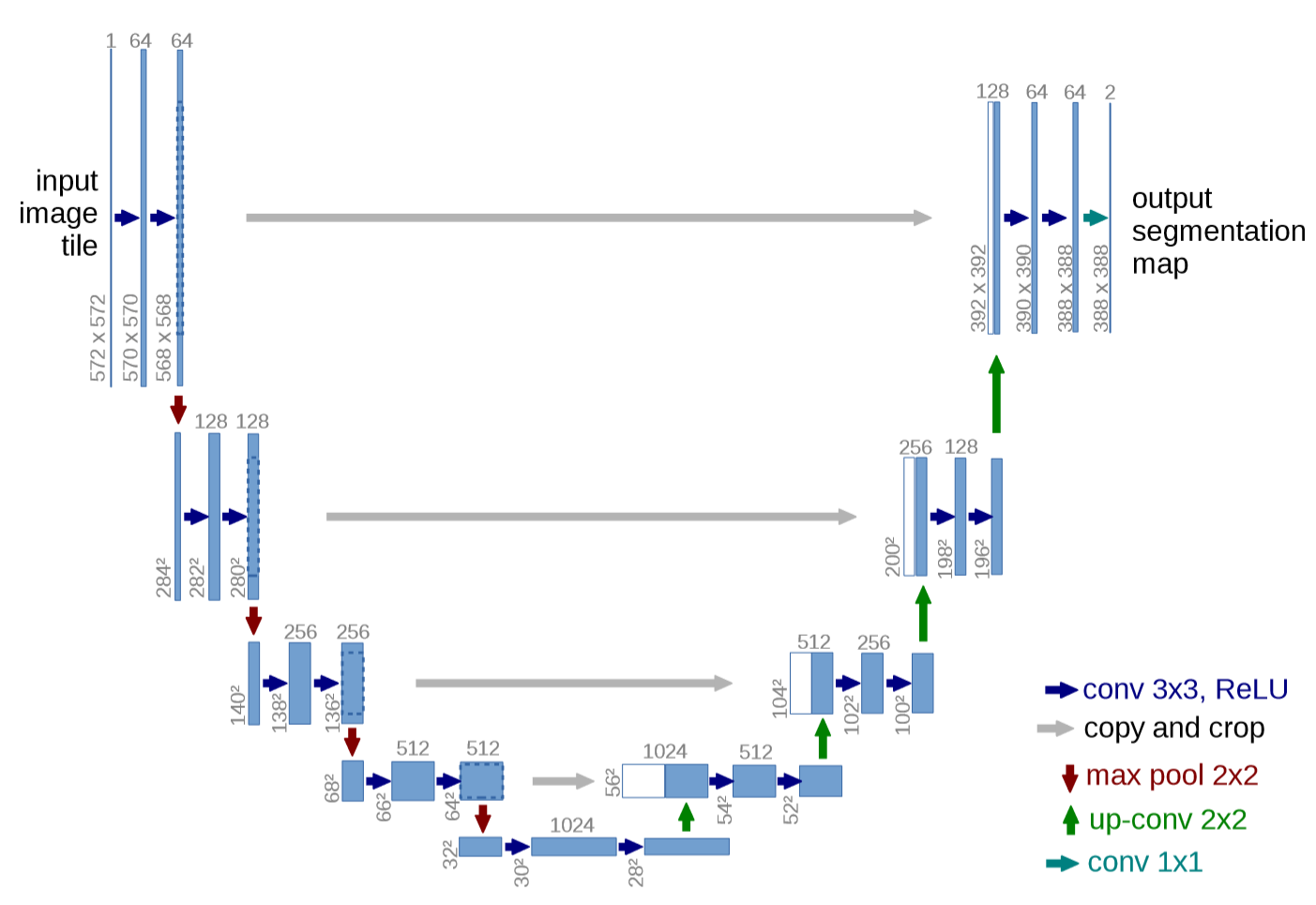
U-Net架构图
Swin Transformer 算法优化操作
Transformer是一种序列到序列的预测体系结构,不需要任何卷积算子,仅依靠自关注机制提取图像特征信息,建立有效的远程依赖关系,Transformer擅长建模全局上下文,但它们在捕获细粒度细节方面存在局限性,特别是对于医学图像,在建模局部信息时缺乏空间诱导偏差,而CNN架构可以很好地弥补这一限制。近年来,随着Transformer在计算机视觉领域的崭露头角,许多研究者将其引入到医学分割领域,并取得了良好的效果,这也为U-Net的优化提供了额外的解决方案。 Swin Transformer 是一种包含滑窗操作,且采用层级设计的视觉 Transformer结构。Swin Transformer 整体架构参考了卷积神经网络的设计方式,设置了不同的阶段,通过补丁合并的方式减小尺寸,来获取不同尺寸的各阶段特征。Swin Transformer 将注意力操作的相关计算约束在每一个窗口内,大幅降低了 Transformer 使用时的计算量。
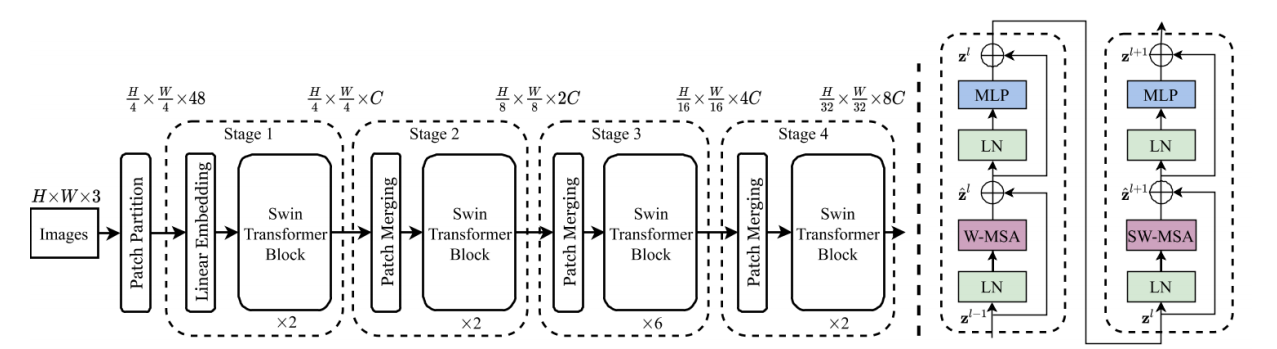
Swin Transformer网络结构图
TGDAUNet 医学图像分割算法网络结构
本项目的TGDAUNet算法网络结构图如右图所示,双支路骨干网络——res2net和SwinTransformer作为特征提取器。多尺度融合(MF)模块基于从CNN骨干网中提取的高级特征信息进行多尺度融合。融合后的特征信息通过PSA模块传递,减少冗余信息的影响。随后,利用Transformer主干网络提取的特征信息建立多尺度长程依赖关系。然后,反向图推理融合(RGF)为边界非重要特征和区域构建图推理,捕获边界线索并使用特征聚合(FA)模块构建全局上下文指导。
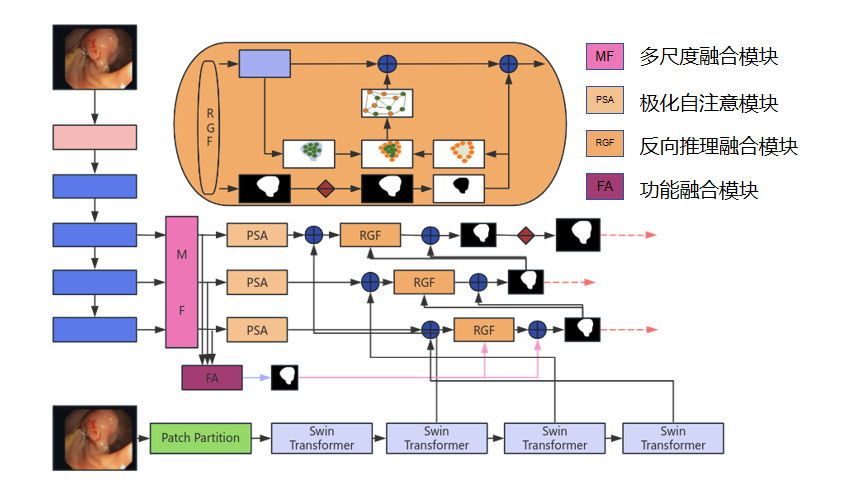
TGDAUNet算法网络结构图
TGDAUNet算法 训练设置和评价指标
在对医学影像模型进行性能评估时,需要借助一些相关的性能指标来判断模型分割结果的优劣。在本项目中,主要使用Loss、Dice、IOU、BAcc四个指标,作为衡量模型优劣。 Loss又称之为交叉熵损失,是一种基于分布的损失函数,最早用于分类任务的损失构建。医学图像分割任务也可以看作为像素级别的分类的任务,可以看作为二分类任务,因此可以使用二元交叉熵损失。Dice系数用来衡量集合间的相似度,表示两个样本之间的相似程度,常用来评估医学影像分割的准确性,两者取值范围均为[0,1]之间。Dice越接近于1,说明分割效果越好,Loss越接近于0,也可以说明分割效果越好。当两个样本相似度越高时,Dice系数的值就越接近于1,而Loss的值便会越接近于0。 交叉比(Intersection over Union, IoU):IoU 是一种用于衡量对象检测任务中模型预测准确性的指标。它计算模型预测的区域与真实标签之间的重叠程度,通常用于评估对象边界框的精度。IoU 的取值范围在 0 到 1 之间,值越高表示预测的区域与真实标签重叠程度越高。 二元准确率(Binary Accuracy, BAcc):BAcc 是一种用于衡量二元分类任务中模型准确性的指标。它考虑了模型在正类样本和负类样本上的准确率,并对两者取平均值。BAcc 的取值范围也在 0 到 1 之间,值越高表示模型的整体准确性越高。
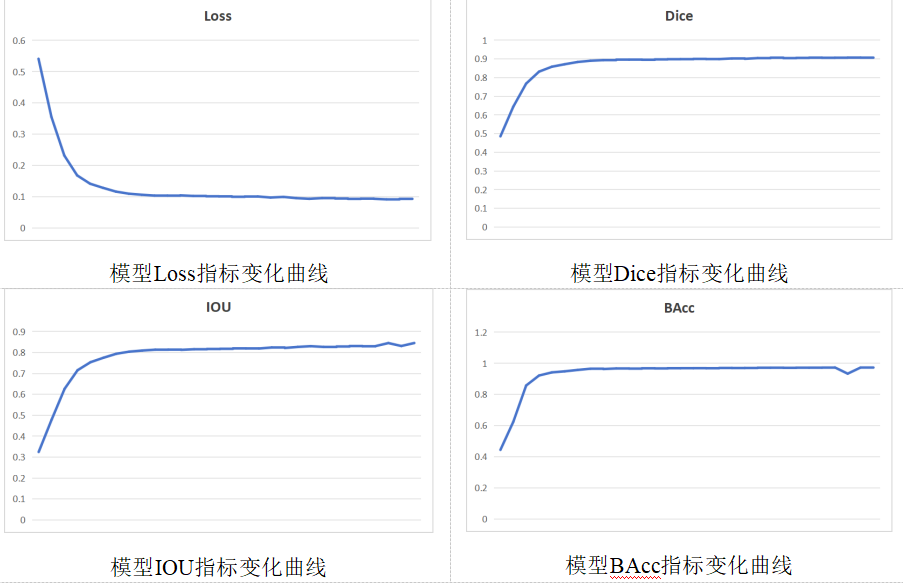
指标变化曲线
数据集及测试展示
结直肠息肉辅助诊断是医学影像领域的一个重要任务,旨在帮助医生准确诊断结直肠息肉病变,并为手术治疗提供指导。为了推动相关算法模型的发展和评估,研究人员开发了一些公开数据集,这些数据集包含了丰富的结直肠影像数据,可供科研机构和学术界使用。通过利用这些数据集,研究人员可以训练和测试各种算法模型,以提高结直肠息肉诊断的准确性和效率,为医生提供更可靠的诊断支持。本项目使用的结直肠息肉诊断数据集如下。 (1)CVC-ClinicDB CVC-ClinicDB数据集是用于结直肠镜图像分析的一个公开数据集,于 2015 年提出,该数据集主要用于结直肠息肉检测任务,即识别结肠镜图像中的息肉。该数据集由从 29 个结肠镜检查视频中提取的 612 个息肉静态图像组成,所有图像的分辨率固定为 384×288 像素。因其同一个片段内的各图像内容是十分相似的,所以对于提高模型的泛化能力较为困难。CVC-ClinicDB 数据集常被研究人员用于模型的测试和验证。 (2) CVC-ColonDB CVC-ColonDB数据集中有 300 个静态样本,每个图像的大小为 574×500 像素,是基于 15 个结肠镜检查视频构建的。该数据集特点在于它包括了不同程度病变的结直肠息肉,从轻微的变形到明显的肿瘤,这使得研究人员能够对不同类型和不同程度的息肉进行研究和评估,有利于提高模型的泛化能力。CVC-ColonDB 已被广泛用于结直肠息肉辅助诊断算法的评估和比较。 (3) Kvasir-SEG Kvasir-SEG数据集于 2020 年提出,是息肉分割领域一个常用的数据集。Kvasir-SEG 数据集由1000 张息肉图像及其相应的掩码组成,样本的分辨率在 332×487 到 1920×1072 像素之间。因其每个图像都配有对应的像素级别标注,更有利于模型进行精细化分割,这对深度学习中的图像分割任务是关键的。并且数据集中包含的多样性图像有助于提高 CNN 模型的泛化能力。
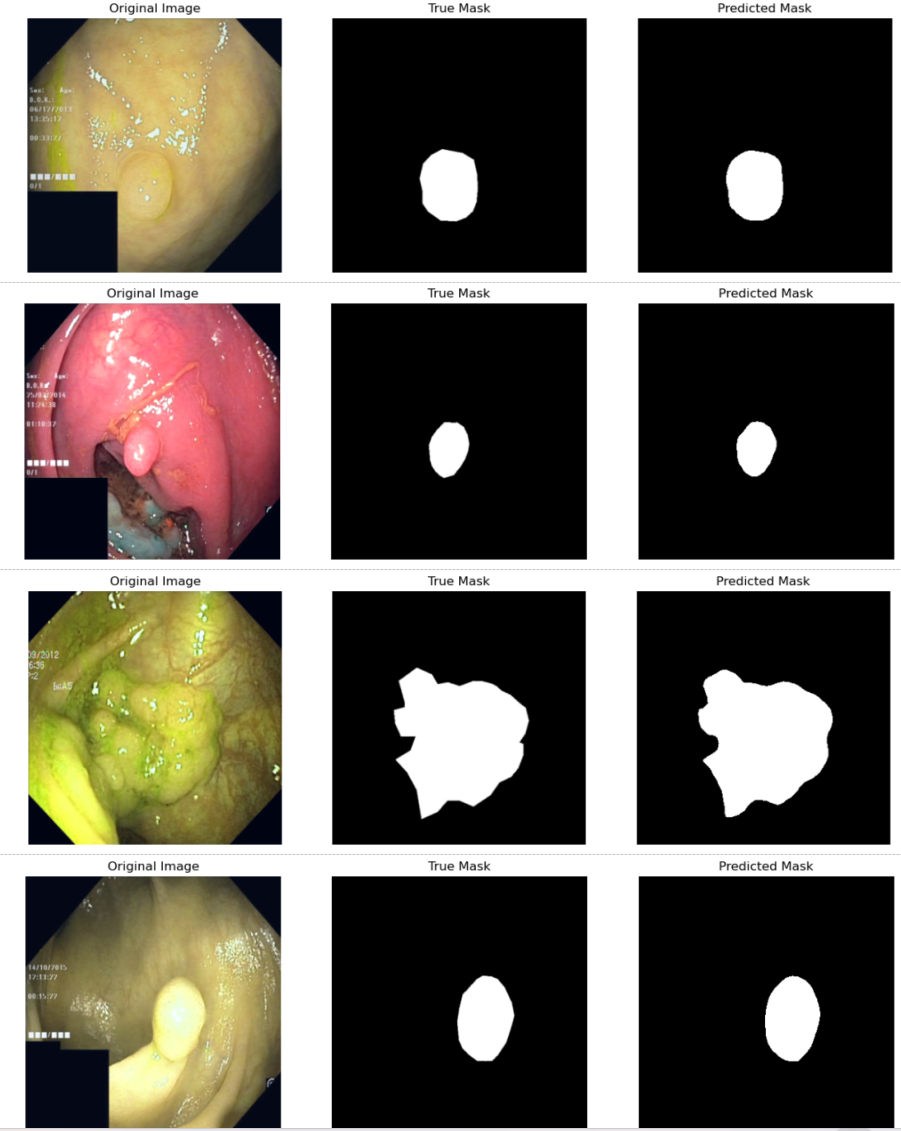
部分测试展示